Parutions
![]()
Les cellules eucaryotes se déplacent sur des surfaces solides grâce à leur cytosquelette d’actine en assemblant localement des foyers d’adhésion focale où le couplage entre l’actine, des molécules d’adhésion et des moteurs moléculaires, les myosines, crée des forces de traction. Des chercheurs du Laboratoire de chimie bactérienne, de l’Institut de pharmacologie moléculaire et cellulaire, et du Max Planck Institute for terrestrial microbiology (Marburg, Allemagne), révèlent que ce type de régulation existe également chez les bactéries en démontrant que l’appareil de motilité de Myxococcus xanthus est activé au pôle avant des cellules ou il recrute le cystoquelette d’actine et désassemblé au pôle arrière. Cette étude est publiée dans la revue The Journal of Cell Biology.
Chez Myxococcus xanthus, une bactérie « sociale », le mouvement sur des surfaces ou motilité, permet de former des structures multicellulaires qui sont des corps fructifères analogues à ceux formés par l’amibe Dictyostelium discoidum. L’assemblage de l’appareil de motilité de Myxococcus, un complexe intégral de l’enveloppe bactérienne fonctionnellement analogue aux complexes d’adhésion focale des eucaryotes, requiert la présence de MglA, une petite protéine de la superfamille Ras. La localisation spécifique sur la face cytoplasmique de l’appareil de motilité de cette protéine est dépendante de la polymérisation de MreB, l’actine bactérienne. Cette régulation est directe car, in vitro, MreB interagit spécifiquement avec la forme GTP de MglA, et non avec la forme associée au GDP.
Afin de tester la régulation de cette interaction, les chercheurs ont observé la motilité de cellules exprimant un variant de MglA qui n’hydrolyse pas le GTP (MglAQ82A). De manière remarquable, ces bactéries sont bloquées dans un mouvement pendulaire « perpétuel » et changent de direction lorsqu’elles ont parcouru une distance correspondant à la taille du corps cellulaire. Ce phénotype est également retrouvé chez des mutants de Plasmodium falciparum, l’agent du paludisme, qui sont incapables de désassembler leur appareil de motilité. Chez Myxococcus xanthus, le complexe de motilité est désassemblé à l’arrière des bactéries, parce que le pôle arrière contient MglB, une GTPase Activating protein (GAP) spécifique de MglA. L’action de MglB active donc la transition MglA-GTP/GDP, ce qui dissocie le complexe de motilité du cytosquelette. Ainsi, chez les mutants MglAQ82A MglB ne peut activer l’hydrolyse du GTP, le complexe reste associé à l’actine et propulse alors la cellule dans la direction opposée. Ce cycle est répété à chaque fois que le complexe atteint l’arrière des bactéries générant le mouvement pendulaire.
Chez les bactéries, le cytosquelette d’actine est généralement impliqué dans la synthèse de la paroi bactérienne, le peptidoglycane. L’exemple de Myxococcus illustre une nouvelle fonction de l’actine bactérienne et constitue un exemple remarquable de convergence évolutive des mécanismes de régulation spatiale du cytosquelette, suggérant que cette bactérie peut être utilisée pour étudier des mécanismes généraux de régulation des complexes de motilité par les protéines G et le cytosquelette d’actine.
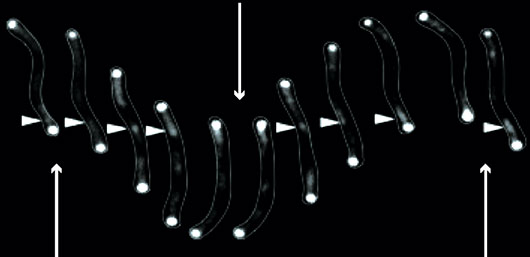
Figure : Motilité pendulaire de Myxococcus xanthus exprimant la protéine de fusion YFP- MglAQ82A bloquée sous sa forme GTP et insensible à l’action de MglB. Une cellule individuelle est représentée à des intervalles de temps de 30 secondes. Un complexe de motilité unique est assemblé au pôle avant de la cellule et garde une position fixe (pointe de flèche blanche), propulsant la cellule. Lorsque le complexe atteint le pôle arrière, il n’est pas désassemblé, ce qui entraine le changement de direction de la cellule (flèche blanche) et le mouvement dans le sens opposé. Le cycle est ainsi répété, entrainant un mouvement pendulaire « perpétuel »
© Tâm Mignot
![]() En savoir plus
En savoir plus
-
The small G-protein MglA connects to the MreB actin cytoskeleton at bacterial focal adhesions.
Anke Treuner-Lange, Eric Macia, Mathilde Guzzo, Edina Hot, Laura Faure, Beata Jakobczak, Leon Espinosa, Damien Alcor, Adrien Ducret, Daniela Keilberg, Jean Philippe Castaing, Sandra Lacas Gervais, Michel Franco, Lotte Søgaard-Andersen, and Tâm Mignot.J Cell Biol. July 13, 2015, doi:10.1083/jcb.201412047
![]() Contact chercheur
Contact chercheur
Tâm Mignot
Laboratoire de Chimie Bactérienne
CNRS UMR 7283, Université Aix Marseille
31, chemin Joseph Aiguier
13009 Marseille
Tél : 04 91 16 45 32